|
Comprehensive discovery and functional characterization of the noncanonical proteome
Chengyu Shi, Fangzhou Liu, Xinwan Su, Zuozhen Yang, Ying Wang, Shanshan Xie, Shaofang Xie, Qiang Sun, Yu Chen, Lingjie Sang, Manman Tan, Linyu Zhu, Kai Lei, Junhong Li, Jiecheng Yang, Zerui Gao, Meng Yu, Xinyi Wang, Junfeng Wang, Jing Chen, Wei Zhuo, Zhaoyuan Fang, Jian Liu, Qingfeng Yan, Dante Neculai, Qiming Sun, Jianzhong Shao, Weiqiang Lin, Wei Liu, Jian Chen, Liangjing Wang, Yang Liu, Xu Li, Tianhua Zhou & Aifu Lin
Cell Research (2025) doi:10.1038/s41422-024-01059-3
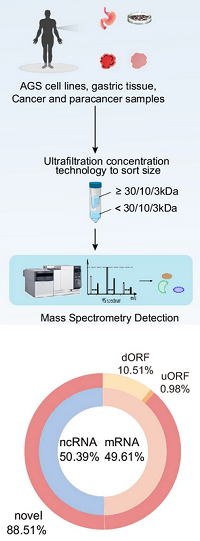
研究人员从Ensembl的人类转录组数据出发,构建了一个包含超过1100万个潜在小开放阅读框(sORFs)的参考数据库。他们对癌症/副癌症组织、正常胃组织和AGS胃癌细胞系的多个样本进行了制备。样本经过研磨和裂解后,使用超滤串联质谱(ultrafiltration tandem MS)进行分析。使用Mascot数据库搜索,研究人员在这1100万个sORFs中发现了8495个与先前未注释的肽段匹配的结果。其中,4866个肽段至少被两个肽段匹配(PSM)支持,2290个肽段在不同样本中都有检测到。
生物信息学分析显示,这些新发现的肽段基因主要位于17、19和22号染色体上。研究人员使用CRISPR筛选技术,识别出1161个影响AGS细胞增殖的肽段候选者,其中大多数似乎作为促增殖调节因子发挥作用。他们还从CRISPR筛选中选取了100个具有显著表型效应的肽段,利用AlphaFold2和肽-蛋白相互作用网络进行了功能预测。最后,研究人员建立了人类新型肽段图谱数据库,以鼓励对这些新发现的肽段进行进一步的表征和验证。这项研究为非经典蛋白质组的全面表征提供了重要信息,强调了这些先前未注释的肽段在胃癌等疾病中的关键作用。
|
|