 |
|
欢迎
Feature驱动的蛋白定量是否比先蛋白鉴定再定量更有效?可能不是,除非你用的是2D 胶的方法。
这个月列举的文章展示了一种揭示蛋白酶体之间相互作用的新方法。如果您最近也有文章发表,并且希望我们列举在下一期的Newsletter中,
请发给我们相关的PDF 或者URL。
这里提醒大家,在接下来的3月22日-3月25日,柏林举办的2015蛋白组学论坛上,我们将开展关于大规模蛋白组学的讨论会。另外我们新增了一个板块:本月Mascot小技巧。
如果你有任何建议和问题,请随时与我们联系。 |
|
|
|
 |
 |
 |
|
先定量后鉴定,还是先鉴定后定量?
许多研究都把蛋白表达量的变化看成是疾病或者应激响应的功能执行者。既然这样,为什么还要浪费时间记录MS/MS谱图,鉴定那些没有变化的蛋白?难道直接关注那些调控的蛋白不是更有效么?
是的,如果你正在使用2D胶,那么那些表达变化的蛋白在胶上肉眼可见,你可以将其切割,消化然后分析。然而,这种方法在如今的shotgun 蛋白组学中无法应用。选择丰度有变化的肽段进行MS/MS 鉴定可能要简单的多,但是一个肽段无法表征一个蛋白丰度的变化。
想要了解更多,请 点击这里。 |
 |
 |
 |
 |
|
使用Mascot发表的优秀文章
这里我们列举了一些最近发表的出色文章,它们使用Mascot进行蛋白鉴定,定量或者特性研究。如果您也想让我们列举您的文章,
请将的PDF 或者URL 发送给我们。
|
|
|
Deciphering Preferential
Interactions within Supramolecular Protein Complexes: the Proteasome Case
Bertrand Fabre, Thomas Lambour, Luc Garrigues, François Amalric, Nathalie Vigneron,
Thomas Menneteau, Alexandre Stella, Bernard Monsarrat, Benoît Van den Eynde, Odile Burlet-Schiltz,
& Marie-Pierre Bousquet-Dubouch
Molecular Systems Biology (2015) 11: 771
蛋白酶体包含一个动态的结构,它们通过改变亚基组成以及与调控颗粒的关系,蛋白间的相互作用来适应生物环境。蛋白酶体如此高水平化的多样性可以反应出每个蛋白酶体特定的作用。
该文章运用label-free定量的蛋白组学方法,描绘了蛋白酶体的异质性。作者利用亲和素纯化,结合高分辨率质谱解析蛋白丰度相关性图谱,分析了亚复合物组成。作者从9株不同的人细胞中,免疫纯化出24个蛋白酶样本,分析了之间的蛋白丰度的关联性,发现两个主要20S 蛋白酶亚型,Sp20S 和iP20S 与不同的调控亚群相互作用。这一方法可以运用到其他大型复合物的研究中。
|
 |
 |
 |
 |
|
2015蛋白组论坛暨大规模蛋白组学论坛
不要错过我们午餐时间的讨论会---Large Scale Proteomics---就在即将到来的
2015蛋白组学论坛
,3月22-25,德国柏林。讨论会将做两场报告,欢迎大家注册。
- Analysis of the human proteome in proteomicsDB, Bernhard Kuster, Technische Universitaet Muenchen
- Reporting complex quantitation data sets using Mascot Insight, Patrick Emery, Matrix Science Ltd.
座位有限,请 尽早注册。 |
 |
 |
 |
 |
|
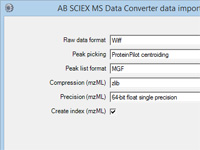
这个月的Mascot 小技巧
您是否知道在新版本Mascot Daemon中,您可以将任何能够处理原始质谱文件的Windows可执行程序添加到Daemon中作为数据导入模块。想要获取更多信息,请在Daemon中按F1 ,更深入的阅读相关的内容:In depth; Data import filters; Adding a new filter.
如果你有任何关于configuration的问题,或者你认为其他Mascot Daemon用户可能想用相同的功能,请发送详情给我们,我们会考虑是否将其添加至标准的configuration中。 |
 |
 |
 |
 |
|
关于 Matrix Science
Matrix Science 为蛋白组学的研究人员以及科学家提供生物信息分析工具,帮助他们更快速,更可信的鉴定和定量蛋白。Mascot 软件全线支持来自AB Sciex,
Agilent, Bruker, Shimadzu, Thermo Scientific 以及 Waters质谱仪生成的质谱数据。
请联系康昱盛以获取更多的信息。
|
 |
 |
|